
Back وصل بديل Arabic Alternativna prerada RNK BS Alternativní splicing Czech Alternatives Spleißen German Alternative splicing English Empalme alternativo Spanish پیرایش دگرسان Persian Splicing alternativo Galician שחבור חליפי HE Alternativno izrezivanje Croatian
|
|
Aquest article o secció necessita millorar una traducció deficient. |
L'empalmament alternatiu (empalmament diferencial) és un procés mitjançant el qual els exons de l'ARN que es produeixen per la transcripció genètica (una transcripció gènica primària o pre-RNAm) es tornen a connectar de moltes maneres durant l'empalmament de l'ARN. Els diferents ARNm resultants poden traduir-se en diferents isoformes de la proteïna. Per tant, un sol gen pot «r múltiples proteïnes.[1]
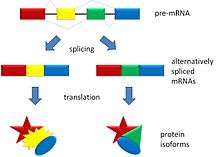
L'empalmament alternatiu es presenta com un fenomen normal en les cèl·lules eucariotes, on s'incrementa en gran manera la diversitat de les proteïnes que poden ser codificades pel genoma.[1] En els éssers humans, un 95% dels gens multiexònics són empalmats alternativament.[2] S'han observat nombroses formes d'empalmament alternatiu, de les quals la més comuna és l'omissió de l'exó. En aquest procés, un exó en particular pot ser inclòs en el ARNm sota certes condicions o en determinats teixits, i omès en l'ARNm en situacions diferents.[1] La producció d'ARNm d'empalmament alternatiu està regulada per proteïnes d'acció trans unides a zones d'acció cis del mateix ARNm. Aquestes proteïnes inclouen activadors d'empalmament que promouen la utilització d'un lloc d'acoblament en particular i repressors d'empalmament que redueixen l'ús d'un altre lloc en particular. Els mecanismes d'empalmament alternatiu són molt variables, i contínuament se'n troben nous exemples, especialment a través de la utilització de tècniques d'alt rendiment. Els investigadors esperen dilucidar plenament els sistemes reguladors que participen en l'empalmament, per poder predir els productes d'empalmament alternatiu d'un determinat gen en unes determinades condicions amb un “codi d'acoblament».[3][4] Les variacions anormals en l'empalmament estan implicades en l'aparició de malalties, ja que són causa d'una gran part dels trastorns genètics humans.[3] També és possible que aquestes variacions anormals en l'empalmament contribueixin al desenvolupament de càncer,[5][6][7] encara que tals productes anormals d'empalmament són generalment salvaguardats i eliminats per un mecanisme de control de qualitat posttranscripcional.[8]
- ↑ 1,0 1,1 1,2 Black, Douglas L. (2003). "Mechanisms of alternative pre-messenger RNA splicing". Annual Reviews of Biochemistry 72 (1): 291–336. doi:10.1146/annurev.biochem.72.121801.161720. PMID: 12626338
- ↑ Pan, Q; Shai O, Lee LJ, Frey BJ, Blencowe BJ (Dec 2008). "Deep surveying of alternative splicing complexity in the human transcriptome by high-throughput sequencing". Nature Genetics 40 (12): 1413–1415. doi:10.1038/ng.259. PMID: 18978789
- ↑ 3,0 3,1 Matlin, AJ; Clark F, Smith, CWJ (May 2005). "Understanding alternative splicing: towards a cellular code". Nature Reviews 6 (5): 386–398. doi:10.1038/nrm1645. PMID: 15956978
- ↑ David, C. J.; Manley, J. L. (2008). "The search for alternative splicing regulators: new approaches offer a path to a splicing code". Genes & Development 22 (3): 279–85. doi:10.1101/gad.1643108. PMC 2731647. PMID: 18245441
- ↑ Skotheim, R I and Nees, M (2007). "Alternative splicing in cancer: noise, functional, or systematic?". The international journal of biochemistry & cell biology 39 (7-8): 1432–49. doi:10.1016/j.biocel.2007.02.016. PMID: 17416541
- ↑ Bauer, Joseph Alan; He, Chunjiang; Zhou, Fang; Zuo, Zhixiang; Cheng, Hanhua; Zhou, Rongjia (2009). Bauer, Joseph Alan. ed. "A Global View of Cancer-Specific Transcript Variants by Subtractive Transcriptome-Wide Analysis". PLoS ONE 4 (3): e4732. Bibcode 2009PLoSO...4.4732H. doi:10.1371/journal.pone.0004732. PMC 2648985. PMID: 19266097
- ↑ Fackenthal, Jd; Godley, La (2008). "Aberrant RNA splicing and its functional consequences in cancer cells" (Free full text). Disease models & mechanisms 1 (1): 37–42. doi:10.1242/dmm.000331. PMC 2561970. PMID: 19048051
- ↑ Danckwardt S, Neu-Yilik G, Thermann R, Frede U, Hentze MW, Kulozik AE (2002). "Abnormally spliced beta-globin mRNAs: a single point mutation generates transcripts sensitive and insensitive to nonsense-mediated mRNA decay". Blood 99 (5): 1811–6. doi:10.1182/blood.V99.5.1811. PMID: 11861299